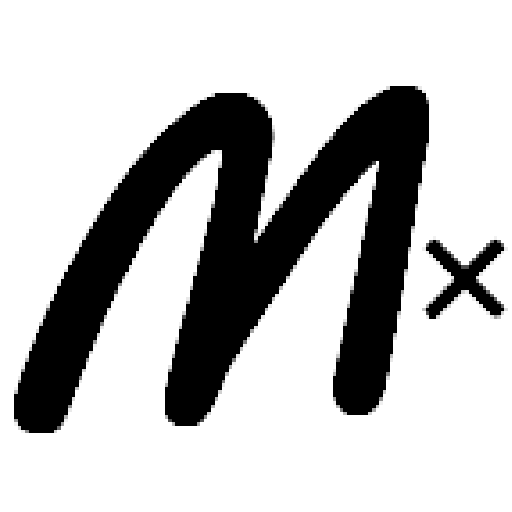準備する試薬類
- PBS: 0.01M Phosphate-Buffered Saline、pH 7.2–7.4
- PBS-Tx: PBS containing 1% (v/v) Triton X-100
- Tris-buffered saline(TBS): 25 mM Tris、140 mM NaCl、3 mM KCl, pH 7.5
- Micro BCA Protein Assay Reagent Kit(Thermo Fisher Scientific Inc., Cat. #23235)
- Cy3 Mono-Reactive Dye Pack 1 mg × 5(GE Healthcare – Life Sciences, Cat. #PA23001)
- Dimethyl formamide(DMF): dehydrated
- Zeba Desalt Spin Columns、0.5 mL (Thermo Fisher Scientific Inc., Cat. #89882)
- Probing Solution(エムックへ問い合わせ、参照論文に組成の記載あり)
- キムテックス・ホワイト(CRECIA、Cat.#63200)
- IgG from human serum (h-IgG) (Sigma-Aldrich, Cat. #I8640)
- Cy3-Streptavidin、1 mg/mL(GE Healthcare – Life Sciences, Cat. #PA43001)
- Mem-PER Plus Membrane Protein Extraction Kit(Thermo Fisher Scientific Inc., Cat. #89842)
- モレキュラーシーブ(Sigma-Aldrich, Cat.#208604)
使用する装置


- 冷却小型遠心機(MX-301: Tomy Seiko Co., Ltd.)
- プレートリーダー(SpectraMax M5: Molecular Devices)
- レクチンマイクロアレイ Ver.1.0 and Ver.2.0(Precision System Science Co., Ltd.、ご不明点があればエムックへ)
- GlycoStation Reader 2300, GlycoSuperLite 2200 (エムック)
- GlycoStation ToolsPro Ver.3.0 (エムック)
- 真空遠心乾燥機(UT-1000, Cat #CVE-200D: Tokyo Rikakikai Co. Ltd. [EYELA])
- インビトロシェーカー(Wave-PR, TAITEC Corp.)
タンパク質の抽出
- Mem-PER Plus Membrane Protein Extraction Kitを使用する
- そのプロトコルについては、Thermofisherの該当ページを参照のこと
タンパク質の定量
- Micro BCA Protein Assay Reagent Kitを使用する
- チューブラックに1.5-mLのマイクロチューブを置き、BSAとラベリングし、240 µL のPBSをアプライする
- 10 µL のBSA 溶液(2 mg/mL)を先のBSAチューブにアプライし、ピペッティングして80 µg/mLのBSA溶液を作る
- BSA(80 µg/mL)チューブと、測定したいサンプルが入ったチューブを氷上におく
- 96-穴MTPに希釈倍率=1/2にて、BSAの希釈系列を作成する。80 µg/mL の濃度から、その1/128倍の濃度まで(溶液量= 100 µL)
- 測定したいサンプルについても、同様な希釈系列を同じ96-穴MTPに作成する
- 2.5 mL の Micro BCA Reagent A(MA)、2.4 mL の Micro BCA Reagent B(MB)そして 100 µL の Micro BCA Reagent C(MC)を15-mL のチューブにアプライし、良く混合する(これをWRと呼ぶことにする)
- 100 µL の WRをすべてのMTPのウェルにアプライする
- プレートシーラーでMTPをシールする
- 37°Cで2時間インキュベートする
- プレートシーラーを剥がす
- 562 nm の吸光度をプレートリーダで測定し、BSAから得られた検量線から測定したいサンプルのタンパク質濃度を計算する
Cy3のラベリング
- タンパク質濃度の測定結果から、サンプルの濃度をPBSをアプライして50 μg/mL に調整する(注意事項 1)
- 100 μg のタンパク質をラベリングするに必要な量に小分けしたCy3が入っているチューブに、20 µL のサンプルをアプライする(注意事項 2)
- ピペッティングで良く混合する
- チューブの側面についている液体を遠心して全て落とす
- チューブをアルミ箔などで遮光し、25°Cにて1時間インキュベートする
フリー Cy3を除去するためのゲルろ過カラム

- Zeba desalt spin columns(0.5 mL)を用意する
- カラムのそこにあるプラグを捩じり取り、キャップを緩める(注意事項 3)
- カラムを2.0 mLのマイクロチューブにはめる
- 1,500 ×g にて1分間遠心し、カラム内の溶液を除去する
- 遠心でレジンは斜めになるが、その斜めになったレジンの頂上部側の位置をカラムにマークする
- 遠心機にカラムをセットする時は、そのマークが常に外側になるようにしてセットする
- 300 μL の TBS をカラムにアプライし、1,500 ×g で1分間遠心し、TBSを除去する
- このステップを合計3回繰り返す
- 新しいサンプル回収用のチューブにカラムをはめ、キャップを除去して、サンプルの全量(20 μL)をレジンの上部側から慎重に注ぎ込む
- サンプルが完全にレジンに吸収された後、25 μL の TBSをスタッカーとしてレジンにアプライする
- 1,500 ×g で2分間遠心し、Cy3がラベリングされたサンプルを回収する
- 使用したカラムは廃棄する
レクチンマイクロアレイにアプライ

- Cy3がラベリングされたサンプル量をピペットで確認する(45μLのはず)
- 455 μL の Probing Solutionをアプライし、500 μLへメスアップする。これによって、タンパク質濃度は、2 μg/mLとなる
- 2,000、1,000、500、250、125、62.5、31.25 ng/mLという7段階の希釈系列をProbing Solutionを用いて作成する(それぞれ、100μL量とする).
- レクチンマイクロアレイを冷凍庫(-20℃)から取り出し、インキュベーションBOXの中に置く
- 8連のマルチピペットを使って、100 μL の Probing Solution をレクチンマイクロアレイのウェルに同時にアプライする
- 数秒間チップを軽く揺すってから、Probing Solutionを捨て、キムテックスの上にチップを裏返して置き、余分なProbing Solutionを吸わせる
- チップを表にして、インキュベーションBOXに置き、チップ表面が乾かない内に、再び100 μL の Probing Solutionをすべてのウェルにアプライする
- これを合計3回繰り返す
- Probing Solutionを捨て、作成した7段階の希釈系列(100 μL)をレクチンマイクロアレイの7つあるウェルに素早くアプライする
- インキュベーションBOXの底に純水を少々入れ、レクチンマイクロアレイを並べてから蓋をして完全にシールし、アルミ箔をかぶせて遮光してインビトロシェーカーの上に置き、20°C で16時間インキュベーションを行う
データの取得と解析法
GlycoStation Reader 2300やGlycoSuperLite 2200を用いてレクチンマイクロアレイをサンプル溶液を捨てずにそのままの状態でスキャンする(注意事項 4)。アレイをスキャナーのステージにセットしてスキャンする前に、スライドグラスの裏面を70%エタノールで拭き、汚れを除去することを忘れないようにする。スキャンした蛍光画像(TIFF画像)をGlycoStation ToolsPro Ver.3.0を用いて数値化し、解析を行う。
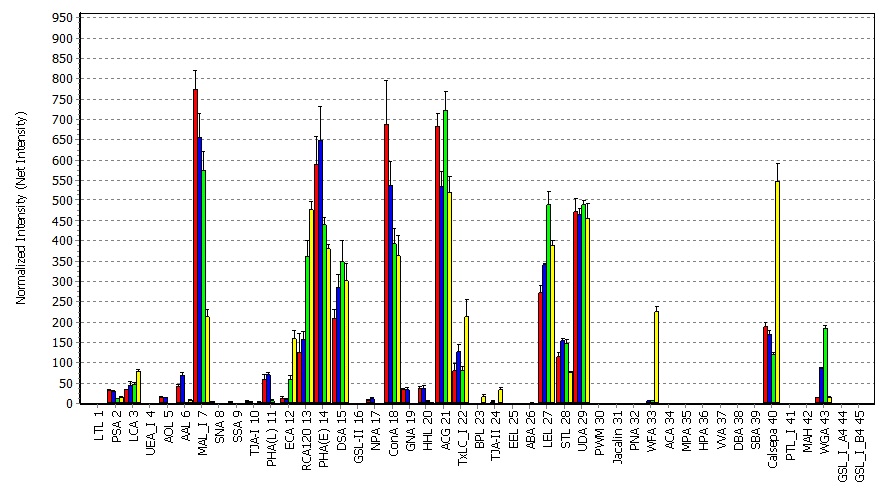
- TIFF画像を読み込んでグリッドを発生させ、スポットの強度を数値化して、Net Intensityを得る(16 bits)
- 2種類のbackground modes(Local Background mode と Global Background mode)を選択することができる
- ゲイン統合(data integrationとの呼ぶ)の機能を使って、飽和しているレクチンの信号を補正し、ダイナミックレンジを広げる。この機能を使うには、異なったゲインで取得した最低二枚の蛍光画像が必要である
- binding curves(濃度が横軸で、縦軸がNet Intensity)を描き、比較解析を行うための最適なタンパク質濃度を選び出す
- 比較糖鎖プロファイリング解析の図を描く(一度に最大10比較を行うことが可能)
- その場合に、幾つかの規格化法を選択することができる(Raw、Marker Intensity、Highest Intensity、Average、特定のレクチンなど)
- 描いた図は、Copy and pasteにて、クリップボードを介してPowerPointらに貼り付けることが出来る
- また、その数値化データは、Excelにエクスポートすることができる
- エクスポートしたデータは、SA/DL Easy(Mx)を使って、統計解析や深層学習をおこなうことが可能である
- Ver3.0では、レクチン名の編集機能を使ってどんなカスタムアレイでも取り扱うことが可能である
抗体オーバーレイ法
- Probing Solutionを用いてサンプルのタンパク質濃度を適当な値に調整する(通常、0.25 µg/mL から 2 µg/mLの範囲)
- 50 µL のサンプルをレクチンマイクロアレイにアプライする
- インキュベータにアレイを並べ、インビトロシェーカーに乗せ、暗所、20°Cにてオーバーナイトでインキュベートする
- human IgG (h-IgG, 10 mg/mL) を2 µL/wellでブロッカーとして各ウェルにアプライする(注意事項 5)
- シェーカー上で、20°C で30分インキュベートする
- PBS-Tx にて3回ウェルをウォッシュする(100 µL/well)
- PBS-Tx (48 µL/well)、h-IgG (10 mg/mL) (2 µL/well)、そしてビオチン化抗体を適切な濃度(4 µg/mL ~ 500 ng/mLの範囲)(50 µL/well)にてアプライする
- シェーカー上で、20°C で1時間インキュベートする
- PBS-Tx (100 µL/well)で3回ウェルをウォッシュする
- PBS-Tx (46 µL/well)をアプライし、次にCy3-Streptavidin(50 µg/mL by PBS)(4 µL/well)をアプライする
- 20°C で30分間インキュベートする
- PBS-Tx (100 µL/well)で3回ウォッシュする
- PBS-Tx ( 50 µL/well)をアプライする
- GlycoStation Reader 2300、或いはGlycoSuperLite 2200でスキャンする

- 他の中性のバッファー類を用いることができるが、アミンを含んだもの(例えばTris buffer)は使えない。アミンはCy3-NHS esterと反応し、タンパク質のラベリング効率が下がってしまう
- Cy3(100 µg/tube)の小分けの仕方。モレキュラーシーブを用いてDMFを一晩かけて脱水する。Cy3 Mono-Reactive Dye Pack(1mg)を50 µL の DMFに溶かす。10本のマイクロチューブにCy3-DMFを5 µL づつピペットで取り分ける。真空遠心乾燥機にそのチューブをセットし、遮光して4時間乾燥を行う。DMFが蒸発し切ったチューブにキャップを固くし、遮光したアルミ袋に乾燥剤と共に入れて4°C で保管する
- カラムのキャップは除去してしまわないこと。レジンが乾いてしまい、タンパク質のエルーション量が変わってしまう可能性がある
- ゲイン統合を行えるように、感度を低感度側から高感度側迄幾つか条件を指定してスキャンしておくことが望ましい(5条件程度)
- 抗体オーバーレイ法に関するオリジナル論文を参照(検出に用いる抗体の糖鎖がレクチンと反応するのをブロッキングする必要があります)
付録
クルードサンプルからのターゲットタンパク質の抽出については、Streptavidin coated magnetic beads、ターゲットタンパク質に対するビオチン化抗体、そしてTBS-Tx (TBS + TritonX-100 1% (v/v))を用意します。ビオチン化抗体をstreptavidin coated magnetic beadsに結合させ、ターゲットタンパク質をビオチン化抗体に捕獲させます。そのターゲットタンパク質は、60°C で5分間の熱処理で、ビオチン化抗体から外れ、回収することができます。ビオチンとストレプトアビジンの結合は強いので、この条件では外れません。